Discapacidades asociadas a enfermedades raras bajo el reconocimiento de entidades (NER) y extracción de relaciones (RE)
El reconocimiento de entidades nombradas (NER) y la extracción de relaciones (RE) son dos de las tareas más estudiadas en el procesamiento biomédico del lenguaje natural (PLN). La detección de términos y entidades específicos y las relaciones entre ellos son aspectos clave para el desarrollo de sistemas automáticos más complejos en el campo biomédico.

Andrés Duque, Juan Martínez-Romo y Lourdes Araujo, del Departamento de Lenguajes y Sistemas Informáticos de la UNED, junto a Hermenegildo Fabregat, colaborador externo que realizó su tesis doctoral en la UNED, publican en Journal of Biomedical Informatics la investigación Negation-based transfer learning for improving biomedical Named Entity Recognition and Relation Extraction, donde se exploran técnicas de aprendizaje de transferencia para incorporar información sobre negación en sistemas que realizan NER y RE. Su objetivo principal es analizar en qué medida la detección exitosa de entidades negadas en tareas separadas ayuda en la detección de entidades biomédicas y sus relaciones.
Andrés Duque, profesor del Departamento de Lenguajes y Sistemas Informáticos de la UNED explica las claves de la investigación.
¿Cuál es el objeto de investigación que ha motivado este artículo?
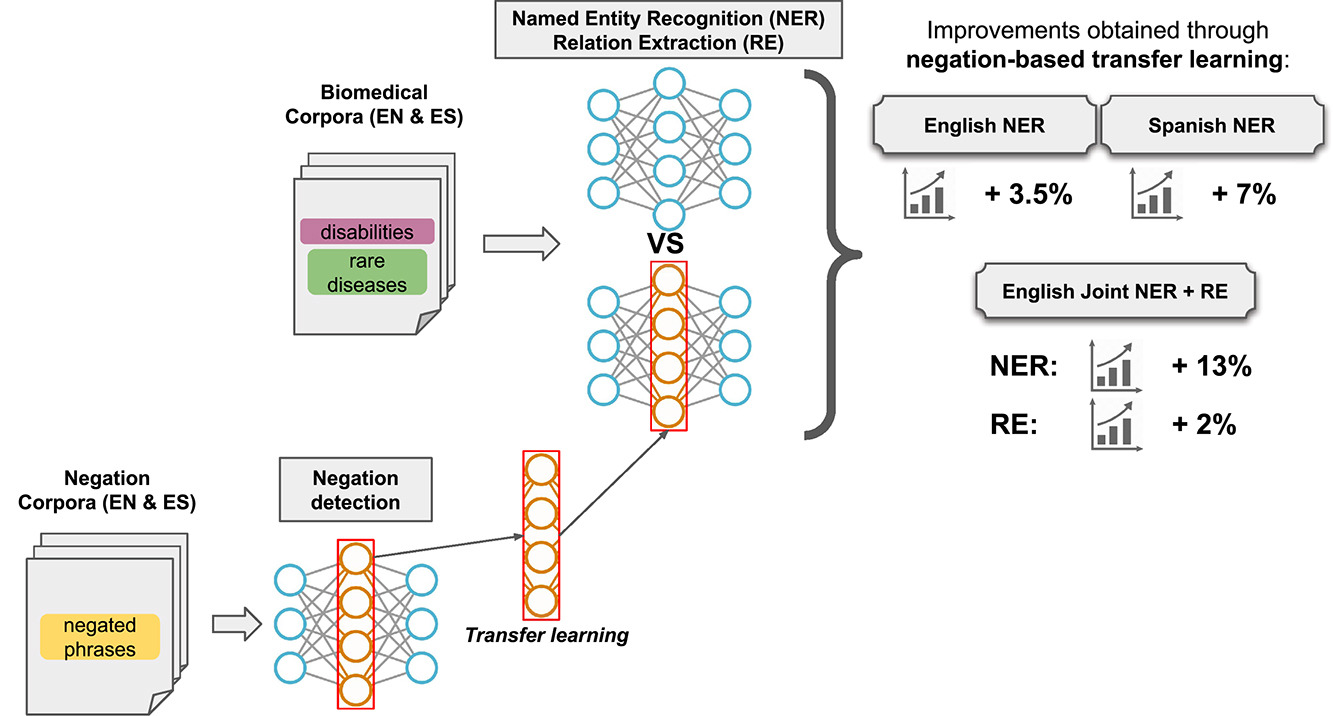
El reconocimiento de entidades nombradas o "Named Entity Recognition" (NER) y la extracción de relaciones entre conceptos o "Relationship Extraction" (RE) son dos de las tareas más importantes dentro del procesamiento del lenguaje natural y la extracción de información en el dominio biomédico. La primera busca encontrar dentro de un texto biomédico (en nuestro caso, resúmenes extraídos de revistas de investigación) determinados conceptos que denominamos entidades, y la segunda tarea se centra en encontrar las relaciones entre este tipo de conceptos previamente encontrados. Para este estudio, nos centramos en la detección de discapacidades y su relación con diferentes enfermedades raras. Este tipo de entidades no están muy estudiadas en las tareas que indicamos, y sin embargo son muy importantes en el dominio biomédico: se estima que un 15% de la población mundial presenta alguna discapacidad, y que entre 300 y 400 millones de personas en el mundo sufre alguna enfermedad rara. Dentro de este contexto, nuestro trabajo se centra en determinar si una correcta detección previa en los textos biomédicos de un fenómeno lingüístico como es la negación es capaz de mejorar aquellos sistemas que se dedican a un posterior reconocimiento de entidades nombradas y a una extracción de relaciones. En nuestra investigación, trabajamos con colecciones de documentos en dos idiomas, español e inglés.
¿Qué supone para el desarrollo de la Medicina esta investigación?
La principal aportación que puede derivarse de nuestra investigación es la aplicación de sistemas previos de detección de la negación en textos biomédicos, para la mejora posterior de otros sistemas que se dediquen al reconocimiento de entidades nombradas y la extracción de relaciones. De esta forma, esto podría repercutir en mejorar el descubrimiento y la extracción de información contenida en textos biomédicos. En la actualidad, la gran cantidad de información dentro del dominio biomédico derivada de la existencia de múltiples fuentes de información e idiomas distintos provoca que un análisis manual de dicha información sea imposible. Por ello, el desarrollo de sistemas automáticos que realicen diversos procesos sobre grandes cantidades de textos del dominio es muy necesario. En concreto, nuestro trabajo está dirigido a la mejora de sistemas que reconozcan nuevas relaciones entre conceptos como pueden ser las discapacidades asociadas a enfermedades raras, que no estén directamente recogidas en la literatura médica actual.
¿Qué les motivó el estudio de esta área de investigación?
La aplicación del Procesamiento del Lenguaje Natural al campo de la medicina es un área de investigación que se ha desarrollado mucho en los últimos años. Como hemos comentado anteriormente, la gran cantidad de recursos textuales no estructurados que se generan en el ámbito de la práctica médica (artículos de investigación, informes médicos, estudios, etc.), suponen un gran desafío a la hora de automatizar cualquier tarea relacionada con el tratamiento de dichos textos. Además, se trata de una oportunidad de aplicar nuestros conocimientos en el área del PLN a un campo como la medicina, en el cual los avances en la investigación se pueden transformar rápidamente en herramientas extremadamente útiles para la sociedad en su conjunto.
¿De qué hipótesis partía y cuáles fueron los resultados?
La principal hipótesis de nuestro trabajo supone que una detección correcta del fenómeno lingüístico de la negación es capaz de aportar un conocimiento útil a un sistema que, a posteriori, pretenda realizar reconocimiento de entidades nombradas y extracción de relaciones sobre textos biomédicos. Para probar esta hipótesis, entrenamos un sistema de aprendizaje profundo basado en redes neuronales para detectar tanto los disparadores (triggers) relacionados con la negación como el ámbito (scope) de dicha negación. Por ejemplo, en la frase ‘casi no tiene accesos de tos, ni presenta fiebre’, un disparador de negación sería ‘casi no’ y el ámbito de esa negación sería ‘casi no tiene accesos de tos’. Un segundo disparador sería ‘ni’, y un segundo ámbito sería ‘ni presenta fiebre’.
Una vez que hemos entrenado este sistema, aplicamos una técnica denominada transfer learning para transferir el conocimiento obtenido por la red neuronal (que se representa en última instancia como una matriz numérica de pesos) a un segundo sistema de aprendizaje profundo, orientado ya a las tareas de reconocimiento de entidades nombradas y extracción de relaciones. De esta forma, podemos comparar si este sistema final obtiene mejores resultados cuando se le inyecta el conocimiento derivado del detector de negación. Los resultados obtenidos nos indican que este entrenamiento previo sobre la negación es capaz de mejorar el reconocimiento de entidades nombradas en un 3.5% en inglés y en un 7% en español, mientras que la tarea de extracción de relaciones se ve mejorada en aproximadamente un 2%.
En su opinión, ¿qué aporta este artículo a lo que ya se había publicado sobre este asunto?
Como hemos comentado, las entidades que se han tratado en este trabajo (discapacidades y enfermedades raras), aunque muy importantes en el ámbito biomédico, no se suelen considerar en tareas clásicas de reconocimiento de entidades o extracción de relaciones, por lo que este trabajo, y más en particular los conjuntos de datos utilizados para su desarrollo pueden significar un punto de partida importante para el desarrollo de nuevos sistemas que aborden este tipo de entidades. Por otro lado, el hecho de presentar una investigación en documentos escritos tanto en inglés como en español supone un importante avance en lo que se refiere al multilingüismo en este dominio, en el que las principales investigaciones se desarrollan principalmente en inglés. Finalmente, hasta donde alcanza nuestro conocimiento sobre el estado del arte en este dominio, no existen trabajos previos que exploren directamente la aplicación de técnicas de transfer learning para incorporar información (y más concretamente, información sobre negación) a sistemas que realizan reconocimiento de entidades nombradas y extracción de relaciones.
Nos gustaría conocer qué ha supuesto para su equipo la publicación de este trabajo en la revisa Journal of Biomedical Informatics.
El equipo UNED que firma este trabajo se compone de un colaborador externo que realizó su tesis doctoral en la UNED, Hermenegildo Fabregat; un profesor contratado doctor, Andrés Duque; un profesor titular de universidad, Juan Martínez-Romo; y una catedrática de universidad, Lourdes Araujo. La publicación del presente trabajo, por una parte, supone un hito importante dentro del proyecto en el que se engloba la investigación realizada. Este proyecto se denomina DOTT-HEALTH (Development Of Text-based Technology to support diagnosis, prevention and HEALTH institutions management), y se lleva a cabo en conjunto con la Universidad del País Vasco y la Universidad Politécnica de Cataluña. En concreto, el subproyecto que lleva a cabo la UNED se denomina INDICA-MED (Information Discovery and Categorization based on language processing for the Medical domain). Por otro lado, la publicación de este artículo se produce en una revista como Journal of Biomedical Informatics, que se encuentra dentro del primer cuartil del ranking "Journal Citation Report" (JCR) en las categorías "Computer Science, Interdisciplinary.

Comunicación UNED
C/ Juan del Rosal, 14 - 28040 Madrid
comunicacion@adm.uned.es
uned.es
